Levende cellen gebruiken netwerken van chemische reacties om allerlei functies uit te voeren. Chemici in Nijmegen hebben nu een controleerbaar remsysteem ontwikkeld om dit soort netwerken beter na te bootsen. Een stap op weg naar een moleculaire computer.
Zonder netwerken geen leven. Veel van de cruciale processen in levende cellen, waaronder de energiehuishouding, de biologische klok en het kopiëren van het DNA, worden uitgevoerd door netwerken van aan elkaar gekoppelde chemische reacties.
Chemici gebruiken de netwerken uit levende cellen als voorbeeld om heel eenvoudige netwerkjes na te bouwen in het lab. Voor het maken van nieuwe, ‘slimme’ materialen die reageren op signalen van buitenaf of voor complete synthetische cellen, zijn deze netwerkjes heel interessant. De groep van Wilhelm Huck, hoogleraar fysische organische chemie aan de Radboud Universiteit, heeft nu een moleculair remsysteem ontwikkeld om de snelheid van de verschillende reacties binnen het netwerk beter te reguleren. Dat biedt mogelijkheden om naar programmeerbare reactiesystemen te gaan, ofwel een moleculaire computer.
De nieuwe rem is een combinatie van een enzym en een zogeheten inhibitor. Enzymen zijn speciale eiwitten die als katalysator werken: ze laten een reactie sneller verlopen, maar reageren zelf niet mee. Bij vrijwel alle reactiestappen in een cel zijn enzymen betrokken. Zonder enzym verlopen die reacties niet of heel erg langzaam. Wil je dus de snelheid van een reactie beïnvloeden, dan is het enzym het beste aangrijpingspunt.
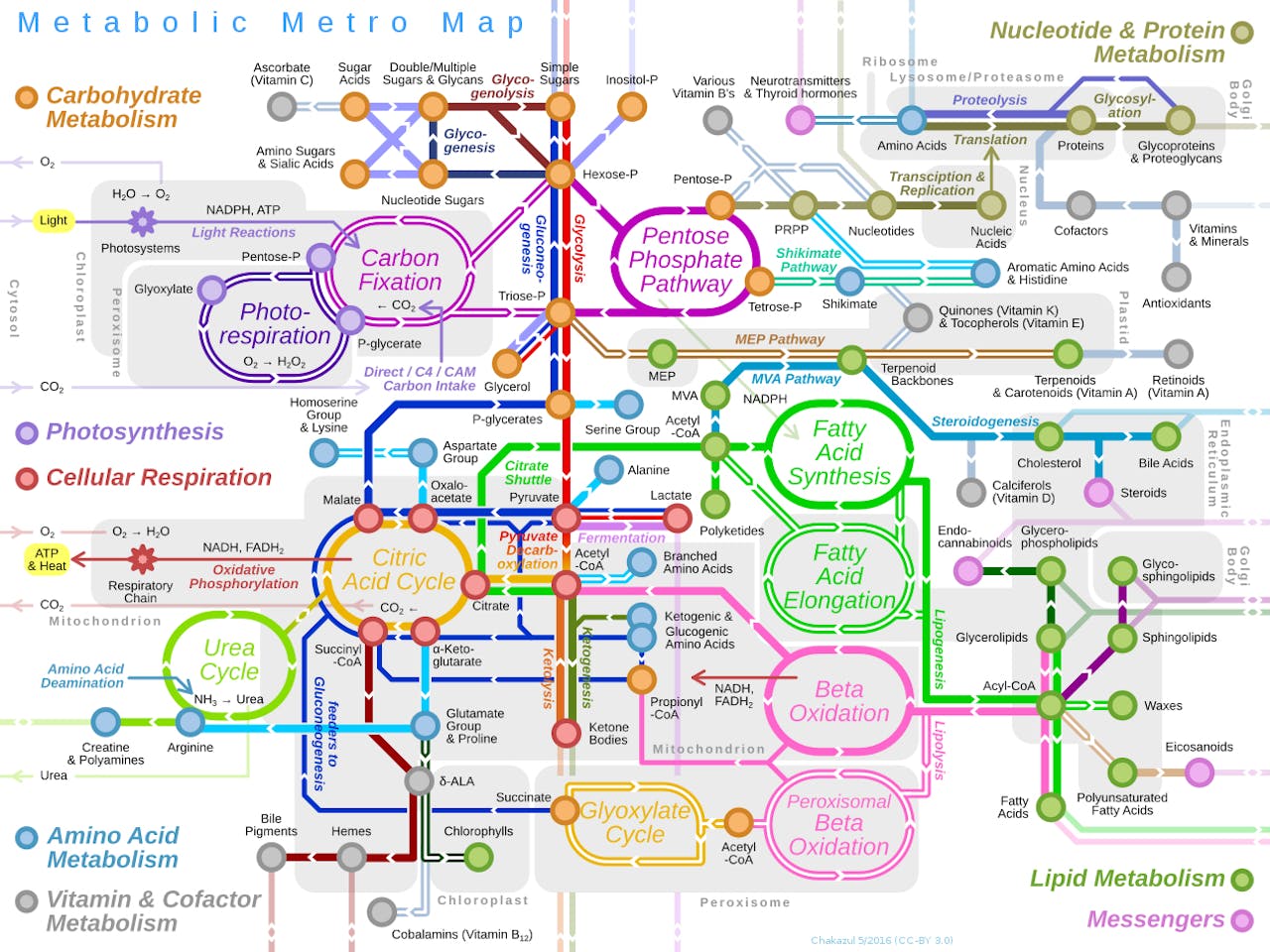
De stofwisseling in een cel afgebeeld als een metrokaart. De kleur van de metrolijn geeft aan welke functie deze route heeft.
Chakazul via Wikimedia Commons CC BY SA 4.0Binden = remmen
Een actief enzym zorgt voor snelheid, maar als je het enzym blokkeert, kan het niet meer werken en heb je in feite een rem. Dat is wat de inhibitor doet. De inhibitor is een specifiek peptide – een klein stukje eiwit – dat gericht bindt aan een specifiek enzym, waardoor dat niet meer z’n rol als katalysator kan vervullen. In hun publicatie in het chemische tijdschrift Angewandte Chemie laten Huck en zijn team drie combinaties zien van een enzym en een inhibitor. Zonder inhibitor voert het enzym op volle kracht zijn taken uit, maar zodra je de inhibitor toevoegt, neemt de activiteit snel af.
Door de hoeveelheid inhibitor te variëren, kun je de activiteit van het enzym en dus de snelheid van de chemische reactie, heel goed beïnvloeden. Het bijzondere van dit systeem is dat het enzym ook langzaam z’n eigen inhibitor kapot kan knippen. Zo ontstaat een dynamisch evenwicht, zegt Huck. “Je hebt inactief enzym met gebonden inhibitor aan de ene kant en vrij, actief enzym met fragmenten van de geknipte inhibitor aan de andere kant. Door inhibitor toe te voegen duw je het evenwicht in de richting van de rem, maar na verloop van tijd is de inhibitor geknipt en wordt het enzym weer actief.”
Verandering levert signaal
De snelheid waarmee de verschillende reacties plaatsvinden, bepaalt voor een belangrijk deel wat het uiteindelijke ‘product’ van het netwerk is. Vaak leiden de reactiestappen in een netwerk tot de productie van een bepaald molecuul dat als signaal dient voor weer andere processen en netwerken in de cel. In levende cellen vinden doorlopend allerlei reacties tegelijkertijd plaats. De productie van een molecuul is op zich geen sterk signaal. Huck: “In een cel gaat het er niet om of een molecuul aanwezig is of niet, maar of de concentratie verandert en hoe snel dat gaat. Dat is de ‘input’ voor de cel om op te reageren.”
Door te spelen met de snelheden van de reacties in een netwerk kun je vanuit hetzelfde startmolecuul (input) verschillende producten (output) leveren. Huck ziet de netwerken die hij bouwt dan ook als programmeerbare systemen. “En de enzymen fungeren hier als de processor: ze verwerken informatie en zetten die om in een actie.”
De drie enzym-inhibitor combinaties die ze nu hebben ontwikkeld, vormen het begin van een veel grotere collectie, aldus Huck. “We laten hiermee het chemische principe zien om aan te tonen dat ons concept werkt. We hebben nog zeker 50 enzymen en 100 inhibitors op ons verlanglijstje staan om verder uit te werken. Dit is allemaal onderdeel van een veel groter plan, namelijk het bouwen van een moleculaire computer.”
Dobberende bolletjes
Lijkt dit op de ‘computers’ op basis van DNA, waar al lang aan wordt gewerkt? “Die leveren ook moleculaire output, namelijk een stuk DNA met de gewenste volgorde, maar die systemen kun je slechts een keer gebruiken, want dan zijn ze op. Ons plan is om de enzymen op te slaan in kleine bolletjes gel die in een reactor ronddobberen. De enzymen vormen als het ware de hardware. De inhibitors vormen de software. Die kun je in allerlei combinaties en verschillende concentraties toevoegen en zo programmeer je het systeem om de gewenste output te leveren.”
Stel dat het lukt, wat gaat die moleculaire computer dan voor ons doen? “Denk aan een systeem dat communiceert met levende cellen. Communicatie tussen cellen verloopt via moleculen, dat zijn de signalen. Cellen scheiden moleculen af en die kunnen als input dienen voor onze computer, waardoor het molecuul wordt omgezet in een stof die bijvoorbeeld een sensor kan detecteren.”
Huck ziet zijn toekomstige computer vooral als een interface tussen bijvoorbeeld een medisch implantaat en levend weefsel, waarbij enerzijds sensoren reageren op, of signalen uitzenden via licht, stroom of pH, en anderzijds levende cellen reageren op moleculen. Je kunt de inhibitors zo ontwerpen dat ze gevoelig zijn voor dergelijke signalen en daarmee van buitenaf aan te sturen zijn. “Deze computer kan signalen van de ene kant omzetten in iets dat voor de andere kant te begrijpen is. Dat biedt allerlei mogelijkheden voor nieuwe materialen of nieuwe productieprocessen.” Voor gedetailleerde toepassingsmogelijkheden is het nog wat vroeg; de moleculaire computer bestaat nog niet. Wanneer kunnen we de eerste demonstratie verwachten? “Over een half jaar ongeveer. We zijn echt al een heel eind op weg.”